Jouez avec vos données de méthylation. Explorez, prédisez, expliquez, le tout sans labo.
EpiXplain est une plateforme de recherche in silico pour tester des hypothèses, composer des signatures CpG/DMR, comparer des modèles de machine learning (11 actuellement), des méthodes de sélection des sondes, et comprendre les prédictions.
Et prochainement : CIIE, moteur contrefactuel CpG « what-if ».
À quoi sert EpiXplain ?
EpiXplain est un environnement d’analyse 100 % in silico pour exploiter des données de méthylation existantes. Il permet de tester rapidement des idées sur vos propres cohortes, en toute traçabilité.
- Charger vos données (CSV/Parquet) et appliquer des modèles en un clic.
- Explorer des signatures DMC/DMR (publiées ou générées) et comparer leurs performances.
- Essayer plusieurs stratégies de **feature selection** (Corrélation, ANOVA, RFImp, PCA, Boruta).
- Identifier les CpG clés d’une prédiction et explorer le contexte génomique (gènes, TFBS, eQTL, EWAS Atlas).
- Vos règles : modèles par défaut par pathologie ou exploration totale.
- Traçable : versions de packages, configs et empreintes à chaque run.
- Simuler bientôt l’impact de modifications CpG (CIIE - moteur contrefactuel).
Pas de wetlab : EpiXplain n’effectue aucune mesure biologique. Il analyse et valorise des données déjà disponibles.
Ce qui fait la différence
Pensé pour la méthylation : matrices ß à grande dimension, DMR/DMC unifiés, recalculs incrémentaux, traçabilité SQL totale. Le futur module CIIE offrira une simulation « what-if » CpG/DMR actionnable et reproductible — une première dans ce domaine.
Statut actuel (honnête)
Data & SQL
- OModèle relationnel robuste (sondes, localisations, annotations, modèles, résultats), versioning et intégrité.
- OEnrichissements biologiques multi-sources (GENCODE, Orphanet, STRING, EWAS, ReMap, FANTOM) unifiés.
- OImport IDAT bout-en-bout : en préparation (aujourd’hui : CSV/Parquet).
Analyses
- OPipeline DMR opérationnel (bump-hunting), export SQL, rattachement DMR?CpG.
- OSélection de variables & modèles (ANOVA, PCA, RFImp, Boruta; RF, XGB, SVM, NB/PLR, MLP).
- OInférence par quorum de modèles ; métriques MCC/F1/AUC enregistrées.
- OConfiguration fine de tous les seuils.
Ops & explicabilité
- ORecalcul nocturne, journalisation SQL/CSV, historisation modèles/configs.
- OExport ONNX & service d’inférence : en cours.
- OExplicabilité SHAP intégrée (local/global) : à implémenter.
- OCIIE — moteur contrefactuel CpG/DMR « what-if » : en développement.
Modules
DMR
Régions différentiellement méthylées, traitement par chromosome, export SQL.
LiveSondes & FS
Charger/sélectionner des CpG, FS (Corrélation, ANOVA, RFImp, PCA, Boruta), composer des signatures.
LiveModèles
Guidage par défauts ou exploration totale : modèles, splits, grilles, comparaison avec calibration.
LiveCIIE
Moteur contrefactuel CpG/DMR : neutraliser sites/régions et observer les bascules de décision (contexte génomique).
En développementCas d’usage impressionnants
Inférence d’un syndrome de Kabuki
Un patient inconnu est classé « Kabuki » par le quorum de modèles avec une probabilité élevée. Les sondes contributives sont listées et exportables pour revue scientifique.
Comparaison trans-maladies
Tester un patient contre toutes les maladies disponibles et comparer les probabilités. Une visualisation radar facilite l’interprétation.
Exploration biologique locale
À partir d’une sonde clé, explorer gènes voisins, TFBS (ReMap), eQTL et traits EWAS reliés — pour générer des hypothèses mécanistiques.
Rare diseases
Episignatures and small cohorts: transparent models with counterfactual checks help protein-level hypotheses.
Oncology
Tumor classifiers & early detection panels. Combine DMRs with curated CpG sets and evaluate robustness.
Environment & other
Age/sex/exposure analyses or custom phenotypes. Bring your own signatures and iterate safely.
Combien ça coûte ?
EpiXplain est proposé sous licence de recherche. Le modèle de prix est simple et transparent :
- Licence par utilisateur scientifique : accès aux modules actifs et mises à jour.
- Frais par unité de calcul (p. ex. 1 000 inférences, 100 entraînements, ou 10 000 simulations CIIE).
Des forfaits académiques, institutionnels ou collaboratifs sont disponibles sur demande.
Prochaines semaines
- Export ONNX + service d’inférence (FastAPI) pour exécutions rapides et portables.
- SHAP intégré (local/global) + stockage en base (top-K par inférence).
- CIIE v1 (CpG unitaire & multi) avec rapport d’impact.
- Propagation automatique de la langue entre pages, exports PDF propres.
À moyen terme
- CIIE au niveau DMR + cartes d’impact.
- Import IDAT complet (SeSAMe/Minfi) et pipeline QC.
- Validation externe & calibration (Brier, fiabilité) sur 2–3 pathologies.
- Préprint méthodologique « counterfactual CpG ».
Illustrations
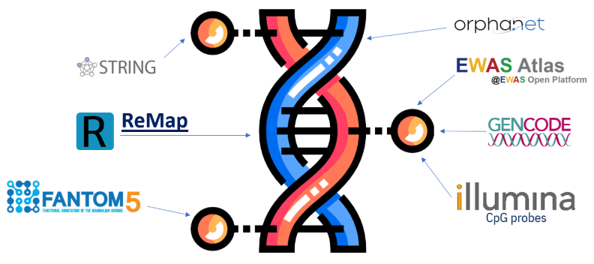
EpiXplain relie automatiquement les sondes CpG à leur contexte biologique grâce à plusieurs bases publiques (ReMap, FANTOM5, EWAS Atlas, GENCODE, Orphanet...). Cette architecture permet d’enrichir les signatures par gènes, régions régulatrices ou fonctions connues.
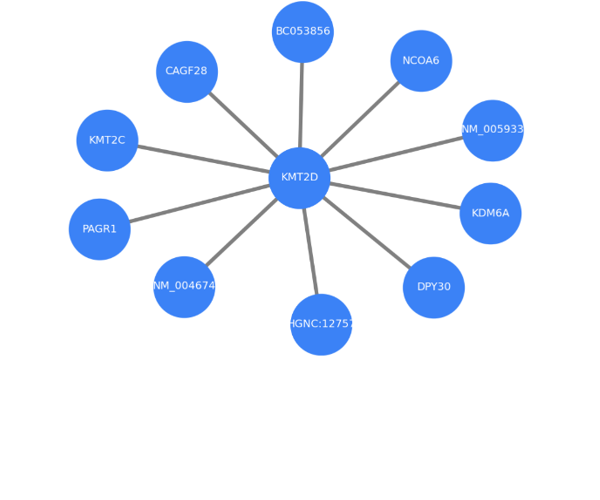
À partir d’un gène pathologique, EpiXplain reconstruit les réseaux d’interaction de premier ordre. Ici, le gène KMT2D (syndrome de Kabuki) est relié à des partenaires impliqués dans des voies de méthylation ou de régulation transcriptionnelle similaires.
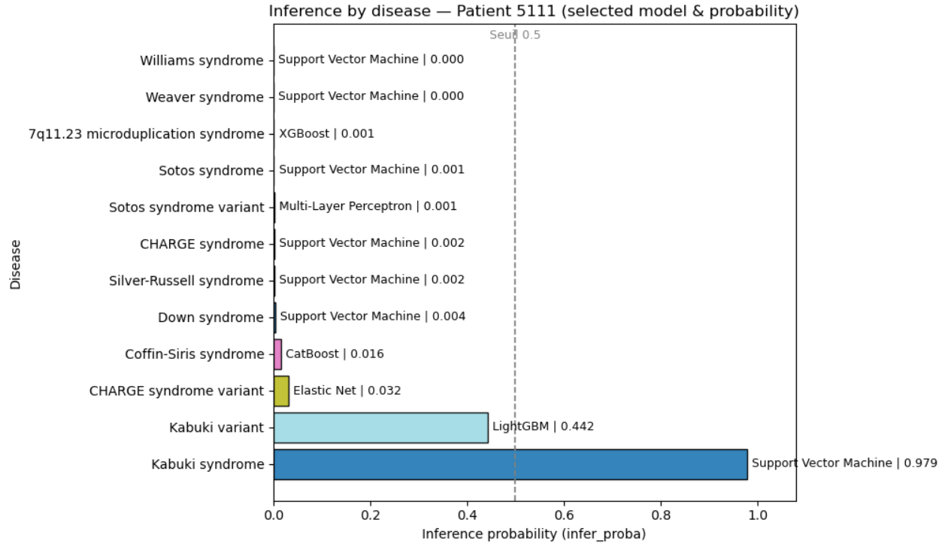
Le modèle d’inférence classe chaque maladie selon sa probabilité. Ici, le patient 5111 est reconnu comme un cas de Kabuki syndrome avec une probabilité de 0.979 (Support Vector Machine), tandis que les autres pathologies obtiennent des scores proches de 0.
Nous contacter
Pilotes (labo, hôpital, groupe de recherche) ou accès anticipé ? Écrivez-nous.
Outil de recherche. Non destiné au diagnostic clinique.
Sécurité & confidentialité
Landing statique (OVH) ; backend applicatif sur infrastructure on-prem sécurisée via VPN. Tous les calculs sont journalisés et versionnés pour la reproductibilité.